南湖新闻网讯(通讯员 黄宇虹)近日,我校作物遗传改良全国重点实验室、湖北洪山实验室小麦团队苏汉东教授课题组在Molecular Plant在线发表“Evolutionary innovations and genetic diversity in angiosperm centromeres”的研究论文。该研究依托最新的T2T基因组测序技术,整合上千份群体基因组和近缘物种数据,从物种内、物种间以及属间多个尺度,对被子植物着丝粒展开了迄今最系统的比较研究,揭示了植物着丝粒在进化、结构与功能之间的内在规律。着丝粒的系统解码也为植物功能着丝粒的理性设计和合成基因组研究奠定了的基础。
染色体是遗传信息的载体,而着丝粒则是染色体上确保细胞分裂准确进行的关键区域,被形象地称为染色体的“指挥中心”。在有丝分裂和减数分裂过程中,着丝粒负责连接纺锤丝,保证染色体被均等分配到子细胞中,对生命的延续至关重要。然而,着丝粒一直以来被认为是基因组中最“混乱”的区域之一。其DNA序列高度重复,在不同物种之间变化极快,却又稳定地承担着不可替代的生命功能。这一“序列高度多变、功能却高度保守”的矛盾现象,构成了困扰科学界数十年的经典难题:为什么着丝粒序列可以不断变化,却始终稳定工作?
为了回答这一问题,研究团队整合分析了17种被子植物400多个完整解析的着丝粒,同时结合上千个泛基因组和近缘物种的群体基因组数据,首次在单碱基精度上,跨越约1.8亿年的演化尺度,系统描绘了植物着丝粒在物种进化、群体分化和染色体重排过程中的动态变化规律。研究发现,被子植物的着丝粒并非结构单一,而是由卫星重复序列和转座子两类重复DNA以不同比例组合而成。不同谱系植物在长期进化过程中形成了各具特色的着丝粒结构模式:有的以高度同质化的卫星序列为主,结构相对简单而稳定;有的则富集大量转座子,形成更为复杂、多层次的着丝粒架构。这种差异反映了不同植物基因组在演化过程中,对结构稳定性与序列创新间作出的不同权衡。进一步分析表明,转座子的插入和扩增是推动着丝粒结构多样化的重要动力。转座子不仅会打断原有的卫星序列结构,还可能改变着丝粒蛋白CENH3的结合区域,从而引发着丝粒大小变化,甚至导致染色体上的位置发生迁移。例如,在某些转座子富集的植物中,着丝粒发生重新定位的频率明显升高,提示转座子活动可能为染色体结构创新提供了重要“原材料”。
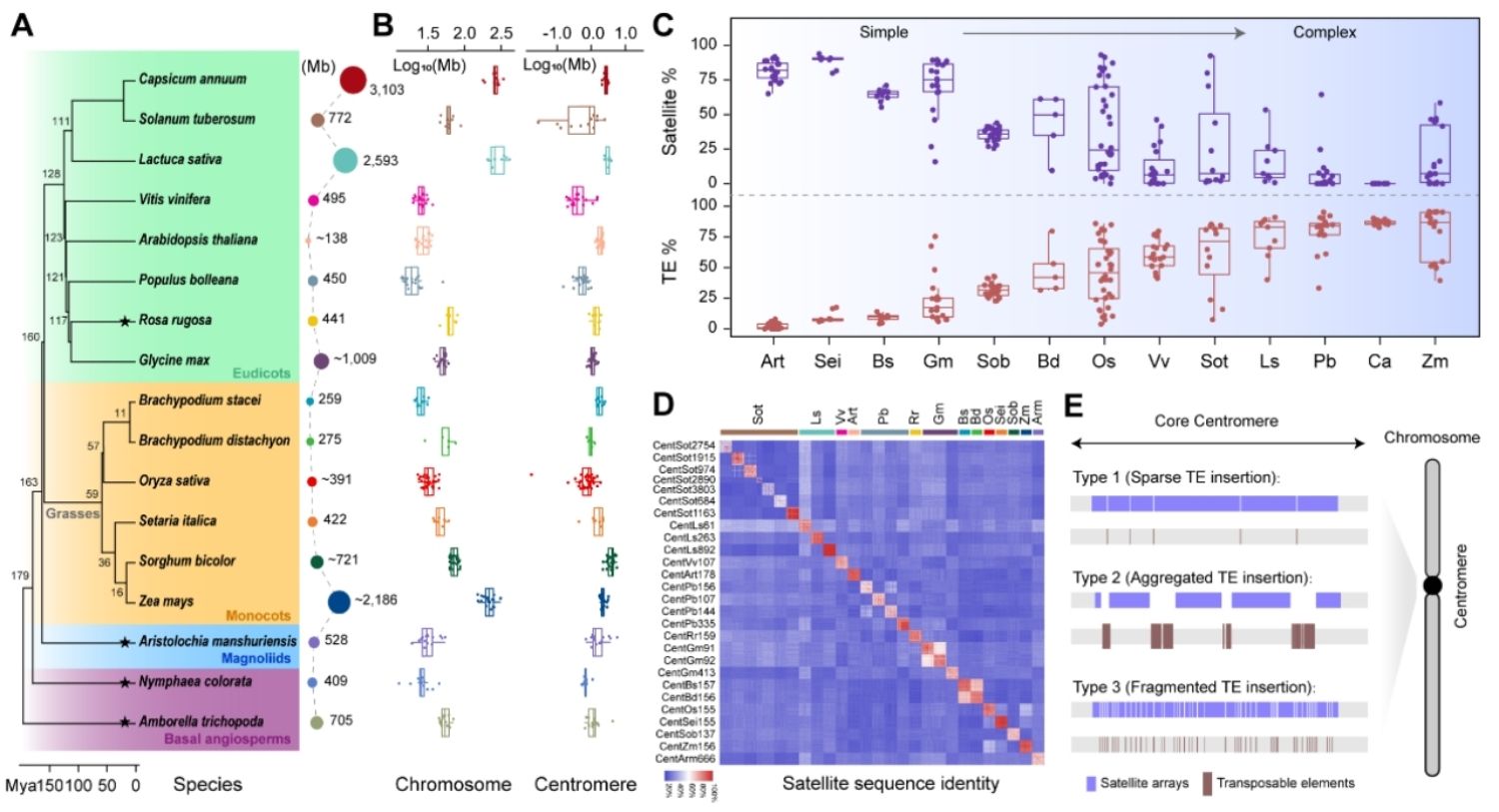
图1 跨植物物种着丝粒卫星重复序列和转座子的系统比较
在群体层面,研究人员观察到,即便在同一物种内部,不同材料或个体之间的着丝粒序列和结构也存在显著差异。其中,卫星重复序列表现出极高的演化活跃性,是基因组中变化最快的区域之一。这一发现打破了“着丝粒是高度保守区域”的传统认知,表明着丝粒更像是一个持续发生序列更新和结构重塑的“进化试验场”。通过对多个植物属的比较分析,研究还揭示了着丝粒的渐进式演化与物种形成之间的潜在联系:近缘物种往往共享相似的着丝粒重复序列,而随着分化时间的延长,这些重复序列会逐渐分化,甚至被全新的类型所取代,并常常伴随染色体重排。这些变化可能在杂交不亲和以及生殖隔离的形成过程中发挥重要作用,为理解植物多样性的起源提供了新的视角。
总体而言,该研究从宏观进化和微观群体变异两个层面,系统描绘了被子植物着丝粒的动态演化图景,提出着丝粒创新是基因组演化、染色体重排与自然选择共同作用的结果。相关成果不仅加深了对染色体与基因组进化规律的理解,也为未来从着丝粒层面精准调控染色体行为、服务作物改良、种质创新和基因组工程研究提供理论支持。
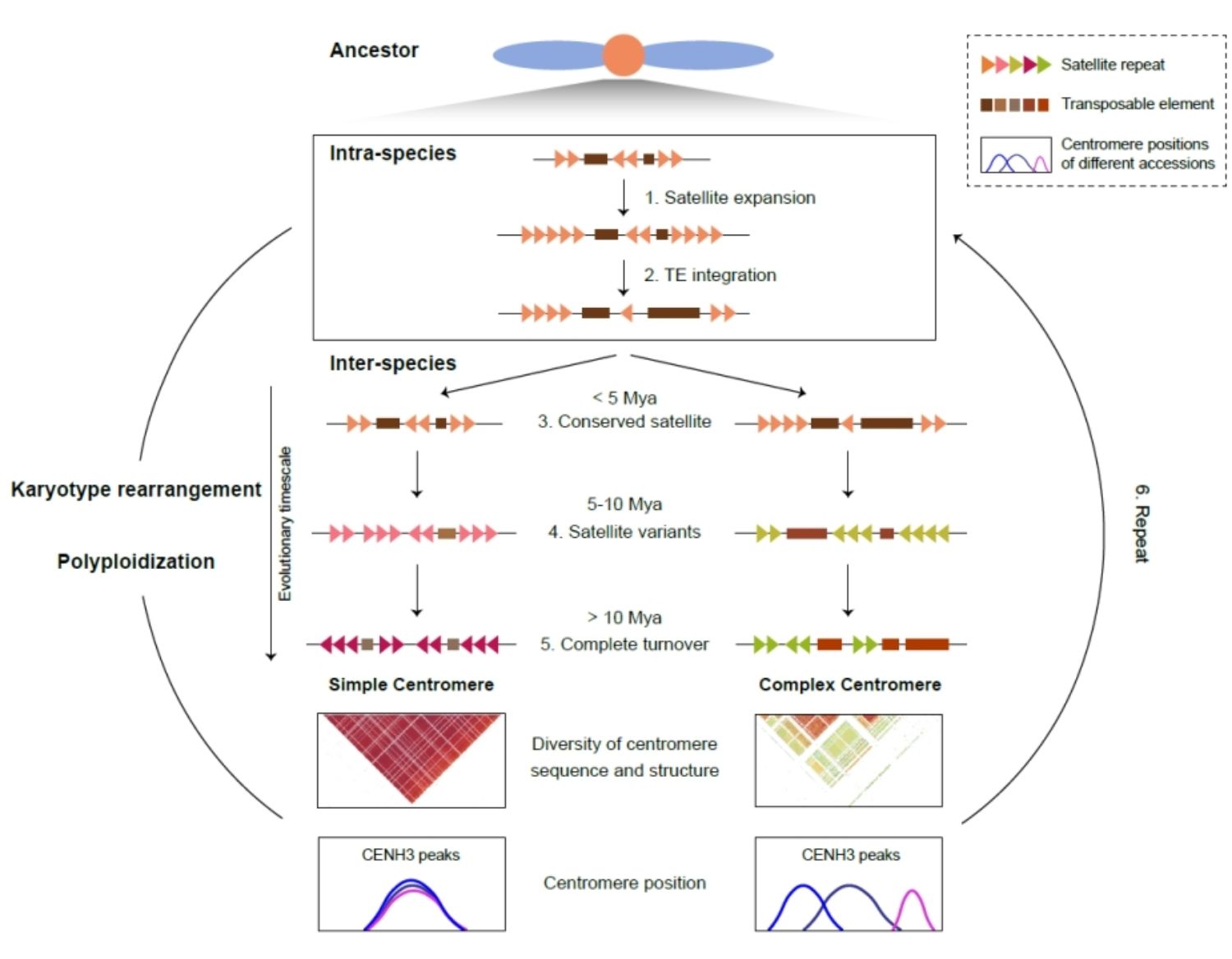
图2 植物种内、种间及属间着丝粒结构与功能演化模式图
开云电竞游戏作物遗传改良全国重点实验室博士后黄宇虹、博士研究生陈川晔、王欣为该论文的共同第一作者,苏汉东为通讯作者,张建伟、马萨里克大学Martin A. Lysak教授参与了部分研究工作。研究得到国家自然科学基金项目和湖北省技术创新计划重点研发项目等重点支持。
审核人:苏汉东
【英文摘要】
Centromeres are indispensable for accurate chromosome segregation, but are subject to rapid sequence turnover while maintaining conserved functions –– a paradox in genome evolution. To unravel this paradox, we integrated over 400 fully resolved centromeres from 17 diploid angiosperms spanning 180 million years of divergence, along with 1,000+ pan-genomic assemblies, resequencing datasets, and congeneric whole-genome sequences. Our study shows that angiosperm centromere organization is determined by lineage-specific combinations of satellite repeat and transposable element (TE), which in turn shape distinct epigenetic landscapes and evolutionary trajectories within centromeres. In particular, TE insertion patterns are found to be one key driver of structural diversification and positional shift of centromeres in angiosperms. Intriguingly, population-level analyses uncovered considerable plasticity in centromere sequences within species, with satellite repeats acting as focal points of evolutionary change and displaying species-specific heterogeneity patterns. Temporal reconstructions across congeneric species revealed the emergence and subsequent differentiation of centromeric repeats, outlining a dynamic continuum from gradual sequence diversification to complete turnover during speciation over time, often accompanied by karyotype reorganization. By integrating intra- and inter-species comparisons, we propose a unifying framework in which centromere innovation is governed by a delicate interplay between genome evolution, chromosomal shuffling and selection constraints, resulting in phylogenomic signatures of centromere-driven speciation.
论文链接:https://www.cell.com/molecular-plant/fulltext/S1674-2052(25)00444-7