南湖新闻网讯(刘昭祥)近日,国际学术期刊Briefings in Bioinformatics在线发表了开云电竞游戏位灯国教授团队与湖北大学付海涛博士的合作研究成果,题为"DistRMI: a deep distance-aware neural network for explainable RNA loop motif-small molecule interaction prediction"。该研究开发了一个集成Transformer和图神经网络(GNN)的深度学习模型——DistRMI,旨在通过引入距离先验信息,实现对RNA环状基序(loop motifs)与小分子相互作用的高精度且可解释的预测。
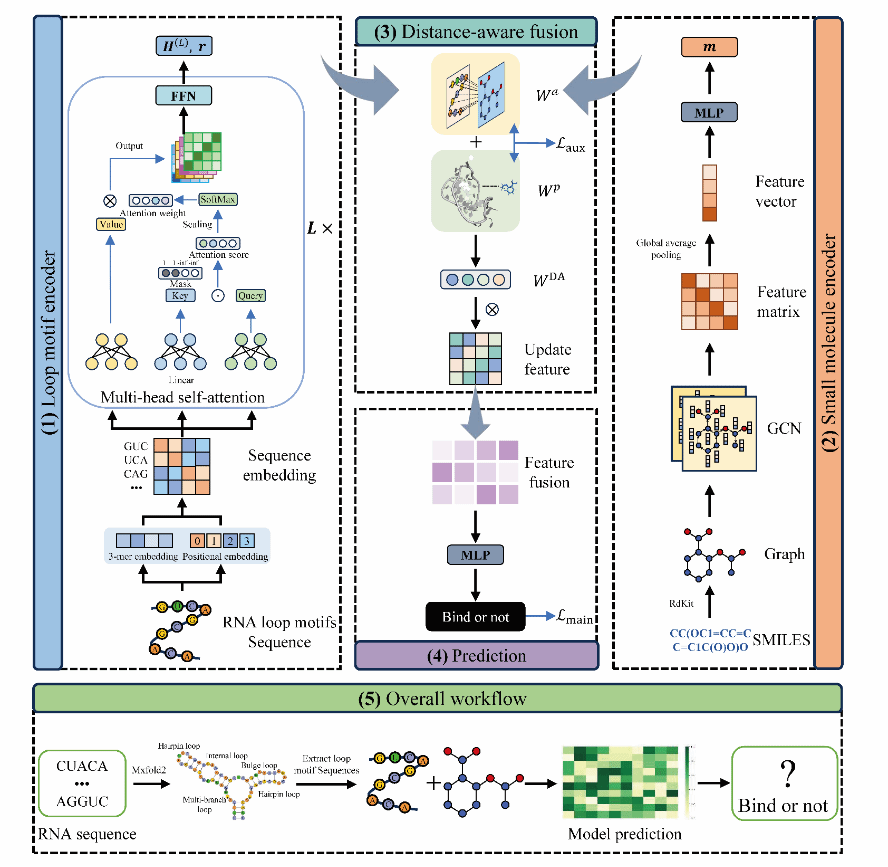
图1. DistRMI模型框架图
RNA作为基因表达的精准调节因子,深度参与了癌症、肌萎缩侧索硬化症及病毒感染等多种疾病的发生与发展。由于其能规避传统蛋白质靶点“不可成药”的局限,RNA已被视为下一代精密治疗的核心方向。然而,当前RNA靶向药物研发面临巨大挑战,核心瓶颈在于湿实验成本高昂且周期冗长。现有的计算方法如分子对接,在处理RNA结构柔性时精度有限;而早期的机器学习模型则往往缺乏透明的决策解释,难以有效引导先导化合物的筛选或阐明深层的分子作用机制。
为突破现有预测工具的局限,研究团队构建了DistRMI框架,通过集成Transformer序列编码器捕捉RNA环状基序特征,并同步利用图卷积网络(GCN)提取小分子的化学拓扑特征。其核心创新在于设计了距离感知融合模块,通过引入碱基与小分子间的3D距离作为空间先验,并利用均方误差(MSE)辅助损失函数约束注意力评分,不仅增强了特征融合的生物学意义,更赋予了模型高度的可解释性。实验结果显示,DistRMI在AUC(93.14%)和AUPR(90.74%)等指标上显著优于Naive Bayes、SVM及RNAmigos等基线模型,且在未知RNA或小分子的“冷启动”场景下依然表现稳健。此外,通过对甲基转移酶核酶(PDB ID: 7V9E_A)等案例的回溯性验证,DistRMI的预测结果与实验观测高度一致,成功识别出关键结合位点。正基于这一卓越的预测精度与机制捕获能力,DistRMI为靶向RNA的药物理性设计提供了透明、可信的决策支撑,有望显著降低药物研发的成本与周期,为攻克重大疾病开辟了新路径。
开云电竞游戏博士研究生刘昭祥为论文的第一作者,开云电竞游戏位灯国教授和湖北大学付海涛博士为共同通讯作者。该项研究得到了国家重点研发计划、国家自然科学基金等多个项目的资助。
审核:位灯国